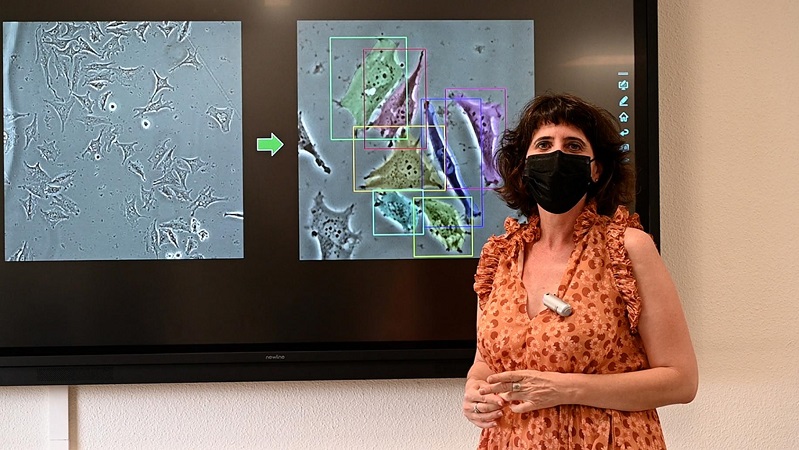
Investigadores del Instituto de Investigación Sanitaria Gregorio Marañón (IiSGM) y de la Universidad Carlos III de Madrid (UC3M), y colaboradores de otras instituciones de Suiza y Suecia, han desarrollado una herramienta, denominada ‘deepImageJ’, que permite procesar fácilmente y analizar imágenes biomédicas (adquiridas por ejemplo, con microscopios o escáneres radiológicos) empleando modelos basados en inteligencia artificial que mejoran la calidad de las imágenes o identifican y clasifican elementos específicos en ellas, entre otras tareas. El trabajo se ha publicado en la revista ‘Nature Methods’.
Los modelos de aprendizaje profundo (deep learning, en inglés) han supuesto un progreso considerable para los muchos campos de investigación e innovación que explotan la información de las imágenes, tales como el diagnóstico clínico por imagen o el desarrollo de nuevos fármacos. En imagen biomédica, por ejemplo, se pueden utilizar para procesar colecciones inmensas de datos, detectar lesiones en tejidos biológicos, identificar sinapsis entre neuronas, y determinar la estructura tanto de la membrana como de los núcleos celulares.
“Durante los últimos cinco años, el procesado de imagen se ha desplazado de los tradicionales métodos matemáticos y basados en la observación hacia el procesamiento guiado por los datos y la inteligencia artificial. Este importante avance favorece que la detección e identificación de datos se realice de forma más sencilla, más rápida y cada vez más automatizada, sobre todo, en el campo de la investigación biomédica. Sin embargo, para utilizar los modelos de aprendizaje profundo se necesitan habilidades de programación que pocos investigadores en ciencias de la vida poseen. Precisamente para facilitar su trabajo, un grupo de expertos en procesado de imagen de diferentes instituciones hemos desarrollado deepImageJ como una herramienta de código abierto que se presenta en un artículo publicado este mes en la revista Nature Methods”, afirma una de las investigadoras principales del proyecto, Arrate Muñoz Barrutia, profesora del Departamento de Bioingeniería e Ingeniería Aeroespacial de la UC3M e investigadora senior del Instituto de Investigación Sanitaria Gregorio Marañón.
Uso de redes neuronales en la investigación biomédica
Este tipo de inteligencia artificial necesita una etapa de entrenamiento que se basa en ejemplos para aprender a realizar una tarea a partir de una cantidad considerable de datos previamente anotados. Funciona de forma similar al software de los sistemas de vigilancia por televisión (en inglés, CCTV systems) que realizan reconocimiento facial, o en los filtros de las aplicaciones de móvil que son capaces de mejorar las fotos.
Los modelos de aprendizaje profundos están basados en arquitecturas computacionales sofisticadas denominadas redes neuronales artificiales. Estas redes están formadas por múltiples capas de procesamiento que pueden modelar los datos a diferentes niveles de abstracción. Como, por ejemplo, para reconocer ciertos tipos de células o lesiones en el tejido o para mejorar la calidad de la imagen.
Inteligencia artificial, pero sin el código
Una vez entrenadas, la información necesaria para realizar la tarea, lo que se denomina modelo computacional de la red neuronal, se almacena en un fichero estructurado en el ordenador que puede ser reutilizado fácilmente con deepImageJ. De hecho, deepImageJ permite que un investigador en cualquier parte del mundo aplique los modelos de aprendizaje profundo entrenados, por ejemplo, por informáticos en España, de forma sencilla.
“Esta herramienta cierra la brecha entre las redes neuronales artificiales y los investigadores biomédicos que ahora pueden pedirle a un informático que diseñe y entrene un algoritmo de aprendizaje automático para llevar a cabo una tarea específica para luego usar el modelo, fácilmente, a través de una interfaz de usuario, sin ver una sola línea de código,” indica Daniel Sage, investigador de la École Polytechnique Fédérale de Lausanne (EPFL Center for Imaging, Suiza) que supervisa el desarrollo del proyecto.
Software colaborativo de código abierto
La herramienta se lanza como un software de código abierto y es gratuito. Está diseñado para ser un recurso colaborativo que permite a ingenieros, informáticos, matemáticos y biólogos trabajar juntos de manera más eficiente. Es decir, investigadores de todo el mundo pueden contribuir a mejorar deepImageJ compartiendo sus experiencias de usuario, así como proponiendo mejoras y requiriendo actualizaciones.
“Nuestro objetivo es que este recurso sea utilizado cada vez por más investigadores desde cualquier ordenador convencional y sin necesidad de tener conocimientos de programación. Además, nuestro grupo de investigación imparte seminarios y desarrolla materiales de formación y recursos online con el objetivo de que los usuarios puedan familiarizarse rápidamente con el nuevo método. Cuantos más usuarios utilicen la herramienta, mayor será la interacción entre los desarrolladores y los investigadores biomédicos. De esta forma se acelerará la difusión de los nuevos desarrollos tecnológicos y, sobre todo, se avanzará en la investigación biomédica”, señala la profesora Arrate Muñoz.
Publicación en Nature Methods
Los autores principales del estudio son Arrate Muñoz Barrutia (UC3M e IISGM) y Daniel Sage (EPFL Center for Imaging). Junto a sus equipos compuestos por Estíbaliz Gómez de Mariscal, Carlos García López de Haro, Michael Unser y Laurène Donati, y colaboradores del Royal Institute of Technology in Stockholm (KTH, Suecia), Wei Ouyang y Emma Lundberg, han conseguido que el proyecto deepImageJ se publique en la revista Nature Methods.
Se trata de una publicación científica mensual revisada que proporciona información relevante sobre nuevas técnicas y métodos de laboratorio. Este trabajo ha recibido apoyo económico del Ministerio de Ciencia, Innovación y Universidades, Agencia Estatal de Investigación, del gobierno español, los fondos FEDER, la acción COST NEUBIAS, una Beca Leonardo 2017 de la Fundación BBVA, EPFL Center for Imaging, Erling-Persson Family Foundation and Kunt and Alice Wallenger Foundation.
Se puede consultar el artículo de la revista Nature Methods en el siguiente enlace https://www.nature.com/articles/s41592-021-01262-9 y acceder a información sobre deepImageJ en la página web https://deepimagej.github.io/deepimagej/index.html